- PUBLICADO:
Proyecto 1000 genomas amplía su oferta de recursos de acceso abierto

Escucha esta noticia
Cargando audio...
Hace algunos años, los creadores del Proyecto 1000 genomas (1000 Genomes Project o 1kGP, en inglés) lanzaron una plataforma de libre acceso, basada en datos de secuenciación del genoma completo (WGS) realizada a 2.504 individuos de 26 poblaciones representativas de los cinco continentes. Este recurso ha sido destacado en numerosas oportunidades por ser el primer esfuerzo a gran escala para ofrecer un catálogo de la variación genética humana.
Pero, hace poco se conoció que la herramienta del Proyecto 1000 genomas ha sido actualizada y expandida, incluyendo a casi todos los tríos de padres e hijos de la colección, junto con las muestras originales. Los datos fueron procesados con alta cobertura utilizando instrumentos Illumina NovaSeq. Tras estos análisis, la plataforma de libre acceso cuenta con 3.202 muestras, incluidos 602 tríos.
“La cohorte del Proyecto 1000 Genomas es un recurso tan valioso que pensamos que sería útil para la comunidad actualizar la secuenciación con la última versión de la tecnología de lectura corta, añadiendo al mismo tiempo la riqueza de las muestras familiares omitidas anteriormente”, explicó el doctor Michael Zody, director científico de biología computacional del NYGC y autor principal del estudio publicado en la revista Cell.
También le puede interesar: Sandoz dejará de ser parte de Novartis en 2023
Claves sobre los procesos de secuenciación genética que ampliaron los datos del Proyecto 1000 genomas
De acuerdo con el artículo, los investigadores utilizaron métodos y algoritmos más avanzados para secuenciar el ADN derivado de las líneas celulares linfoblastoides (LCLs; es decir, células B humanas inmortalizadas de sangre periférica) de la cohorte ampliada hasta una profundidad objetivo de 30X de cobertura del genoma.
Posteriormente, se realizó la identificación de variantes de un solo nucleótido (SNV) y de inserciones y deleciones cortas (INDEL), proceso que consiste en la localización de puntos de variantes, a partir de los datos de la secuencia en relación con la referencia del genoma humano y el genotipado de las muestras de la cohorte. En la actualización del Proyecto 1000 genomas también se incluyó el descubrimiento de un amplio conjunto de variantes estructurales (VS) y su genotipificación, gracias a la integración de múltiples enfoques analíticos.
No olvide leer: FDA autoriza dosis de refuerzo para la variante ómicron de Pfizer y Moderna
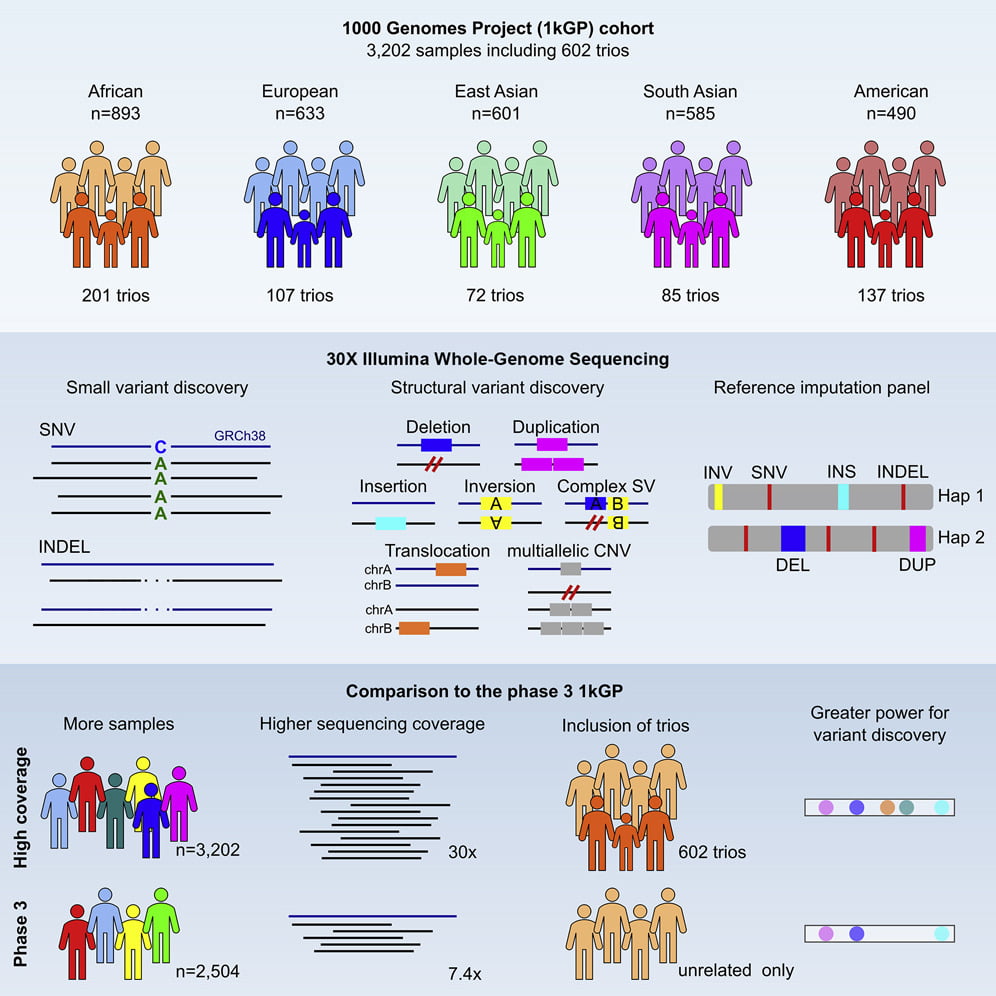
En general, el estudio muestra mejoras significativas tanto en el poder de descubrimiento como en la precisión de las llamadas de variantes, especialmente entre los SNV raros, así como los INDEL y los SV que abarcan el espectro de frecuencias, que antes eran inaccesibles con la secuenciación de baja cobertura, coinciden los investigadores.
Uno de los elementos fundamentales de la plataforma es el panel de referencia para la imputación de variantes. Ahora, la ampliación del recurso original facilitó la actualización de este panel para incluir más variantes descubiertas mediante WGS de alta cobertura y familias de tríos. La información ya ha sucitado el interés de la comunidad científica, ya que la naturaleza de acceso abierto del Proyecto 1000 genomas permite la distribución pública de datos genéticos sin restricciones de uso.
La investigación fue un trabajo colaborativo entre el Centro del Genoma de Nueva York (NYGC) con con grupos del Hospital General de Massachusetts, la Universidad de Yale y el Consorcio de Variación Estructural del Genoma Humano (HGSVC).
Le puede interesar: Supersalud solicitó a las EPS retomar a la vacunación covid-19 como prioridad

























![{"type":"elementor","siteurl":"https://consultorsalud.com/wp-json/","elements":[{"id":"37e6683","elType":"widget","isInner":false,"isLocked":false,"settings":{"text":"IR AL CURSO","link":{"url":"https://formarsalud.com/cursos/estrategias-para-el-saneamiento-financiero-de-ips-en-crisis/","is_external":"","nofollow":"","custom_attributes":""},"align":"center","badge_text":"Badge","button_effect":"c","advanced_button_text_color":"#FFFFFF","button_background_background":"classic","button_border_style":"none","button_hover_background_background":"classic","button_hover_border_style":"none","wcm_element_visibility_content_restricted_message":"You are not authorized to view this content.","ep_widget_cf_colors":"#D30C5C, #0EBCDC, #EAED41, #ED5A78, #DF33DF","ep_display_conditions":[],"ep_parallax_effects_transition_for":"all","ep_parallax_effects_transition_duration":"100","ep_parallax_effects_transition_easing":"linear","__globals__":{"advanced_button_text_color":"","button_background_color":"globals/colors?id=3ab82b4d","button_hover_background_color":"globals/colors?id=50006a26"},"advanced_button_padding":{"unit":"px","top":"7","right":"20","bottom":"7","left":"20","isLinked":false},"advanced_button_padding_tablet":{"unit":"px","top":"7","right":"20","bottom":"7","left":"20","isLinked":false},"ep_widget_cf_shapes":"square|circle","ep_widget_cf_shapes_emoji":"🎃|🎄|💜","ep_widget_cf_shapes_svg":"M167 72c19,-38 37,-56 75,-56 42,0 76,33 76,75 0,76 -76,151 -151,227 -76,-76 -151,-151 -151,-227 0,-42 33,-75 75,-75 38,0 57,18 76,56z|M120 240c-41,14 -91,18 -120,1 29,-10 57,-22 81,-40 -18,2 -37,3 -55,-3 25,-14 48,-30 66,-51 -11,5 -26,8 -45,7 20,-14 40,-30 57,-49 -13,1 -26,2 -38,-1 18,-11 35,-25 51,-43 -13,3 -24,5 -35,6 21,-19 40,-41 53,-67 14,26 32,48 54,67 -11,-1 -23,-3 -35,-6 15,18 32,32 51,43 -13,3 -26,2 -38,1 17,19 36,35 56,49 -19,1 -33,-2 -45,-7 19,21 42,37 67,51 -19,6 -37,5 -56,3 25,18 53,30 82,40 -30,17 -79,13 -120,-1l0 41 -31 0 0 -41z|M449.4 142c-5 0-10 .3-15 1a183 183 0 0 0-66.9-19.1V87.5a17.5 17.5 0 1 0-35 0v36.4a183 183 0 0 0-67 19c-4.9-.6-9.9-1-14.8-1C170.3 142 105 219.6 105 315s65.3 173 145.7 173c5 0 10-.3 14.8-1a184.7 184.7 0 0 0 169 0c4.9.7 9.9 1 14.9 1 80.3 0 145.6-77.6 145.6-173s-65.3-173-145.7-173zm-220 138 27.4-40.4a11.6 11.6 0 0 1 16.4-2.7l54.7 40.3a11.3 11.3 0 0 1-7 20.3H239a11.3 11.3 0 0 1-9.6-17.5zM444 383.8l-43.7 17.5a17.7 17.7 0 0 1-13 0l-37.3-15-37.2 15a17.8 17.8 0 0 1-13 0L256 383.8a17.5 17.5 0 0 1 13-32.6l37.3 15 37.2-15c4.2-1.6 8.8-1.6 13 0l37.3 15 37.2-15a17.5 17.5 0 0 1 13 32.6zm17-86.3h-82a11.3 11.3 0 0 1-6.9-20.4l54.7-40.3a11.6 11.6 0 0 1 16.4 2.8l27.4 40.4a11.3 11.3 0 0 1-9.6 17.5z","add_custom_attributes":"","custom_attributes":"","button_size":"md","onclick":"","onclick_event":"","align_tablet":"","align_mobile":"","button_icon":{"value":"","library":""},"icon_align_choose":"center","icon_align":"right","icon_indent":{"unit":"px","size":8,"sizes":[]},"show_button_badge":"","badge_align":"right","badge_indent":{"unit":"px","size":8,"sizes":[]},"button_css_id":"","attention_button":"","button_background_gradient_notice":"","button_background_color":"","button_background_color_stop":{"unit":"%","size":0,"sizes":[]},"button_background_color_stop_tablet":{"unit":"%"},"button_background_color_stop_mobile":{"unit":"%"},"button_background_color_b":"#f2295b","button_background_color_b_stop":{"unit":"%","size":100,"sizes":[]},"button_background_color_b_stop_tablet":{"unit":"%"},"button_background_color_b_stop_mobile":{"unit":"%"},"button_background_gradient_type":"linear","button_background_gradient_angle":{"unit":"deg","size":180,"sizes":[]},"button_background_gradient_angle_tablet":{"unit":"deg"},"button_background_gradient_angle_mobile":{"unit":"deg"},"button_background_gradient_position":"center center","button_background_image":{"url":"","id":"","size":""},"button_background_image_tablet":{"url":"","id":"","size":""},"button_background_image_mobile":{"url":"","id":"","size":""},"button_background_position":"","button_background_position_tablet":"","button_background_position_mobile":"","button_background_xpos":{"unit":"px","size":0,"sizes":[]},"button_background_xpos_tablet":{"unit":"px","size":0,"sizes":[]},"button_background_xpos_mobile":{"unit":"px","size":0,"sizes":[]},"button_background_ypos":{"unit":"px","size":0,"sizes":[]},"button_background_ypos_tablet":{"unit":"px","size":0,"sizes":[]},"button_background_ypos_mobile":{"unit":"px","size":0,"sizes":[]},"button_background_attachment":"","button_background_repeat":"","button_background_repeat_tablet":"","button_background_repeat_mobile":"","button_background_size":"","button_background_size_tablet":"","button_background_size_mobile":"","button_background_bg_width":{"unit":"%","size":100,"sizes":[]},"button_background_bg_width_tablet":{"unit":"px","size":"","sizes":[]},"button_background_bg_width_mobile":{"unit":"px","size":"","sizes":[]},"button_background_video_link":"","button_background_video_start":"","button_background_video_end":"","button_background_play_once":"","button_background_play_on_mobile":"","button_background_privacy_mode":"","button_background_video_fallback":{"url":"","id":"","size":""},"button_background_slideshow_gallery":[],"button_background_slideshow_loop":"yes","button_background_slideshow_slide_duration":5000,"button_background_slideshow_slide_transition":"fade","button_background_slideshow_transition_duration":500,"button_background_slideshow_background_size":"","button_background_slideshow_background_size_tablet":"","button_background_slideshow_background_size_mobile":"","button_background_slideshow_background_position":"","button_background_slideshow_background_position_tablet":"","button_background_slideshow_background_position_mobile":"","button_background_slideshow_lazyload":"","button_background_slideshow_ken_burns":"","button_background_slideshow_ken_burns_zoom_direction":"in","button_border_width":{"unit":"px","top":3,"right":3,"bottom":3,"left":3,"isLinked":true},"button_border_width_tablet":{"unit":"px","top":"","right":"","bottom":"","left":"","isLinked":true},"button_border_width_mobile":{"unit":"px","top":"","right":"","bottom":"","left":"","isLinked":true},"button_border_color":"#666","advanced_button_radius":{"unit":"px","top":"","right":"","bottom":"","left":"","isLinked":true},"advanced_button_radius_tablet":{"unit":"px","top":"","right":"","bottom":"","left":"","isLinked":true},"advanced_button_radius_mobile":{"unit":"px","top":"","right":"","bottom":"","left":"","isLinked":true},"advanced_button_padding_mobile":{"unit":"px","top":"","right":"","bottom":"","left":"","isLinked":true},"advanced_button_shadow_box_shadow_type":"","advanced_button_shadow_box_shadow":{"horizontal":0,"vertical":0,"blur":10,"spread":0,"color":"rgba(0,0,0,0.5)"},"advanced_button_shadow_box_shadow_position":" ","advanced_button_typography_typography":"","advanced_button_typography_font_family":"","advanced_button_typography_font_size":{"unit":"px","size":"","sizes":[]},"advanced_button_typography_font_size_tablet":{"unit":"px","size":"","sizes":[]},"advanced_button_typography_font_size_mobile":{"unit":"px","size":"","sizes":[]},"advanced_button_typography_font_weight":"","advanced_button_typography_text_transform":"","advanced_button_typography_font_style":"","advanced_button_typography_text_decoration":"","advanced_button_typography_line_height":{"unit":"px","size":"","sizes":[]},"advanced_button_typography_line_height_tablet":{"unit":"em","size":"","sizes":[]},"advanced_button_typography_line_height_mobile":{"unit":"em","size":"","sizes":[]},"advanced_button_typography_letter_spacing":{"unit":"px","size":"","sizes":[]},"advanced_button_typography_letter_spacing_tablet":{"unit":"px","size":"","sizes":[]},"advanced_button_typography_letter_spacing_mobile":{"unit":"px","size":"","sizes":[]},"advanced_button_typography_word_spacing":{"unit":"px","size":"","sizes":[]},"advanced_button_typography_word_spacing_tablet":{"unit":"em","size":"","sizes":[]},"advanced_button_typography_word_spacing_mobile":{"unit":"em","size":"","sizes":[]},"button_width":{"unit":"px","size":"","sizes":[]},"button_width_tablet":{"unit":"px","size":"","sizes":[]},"button_width_mobile":{"unit":"px","size":"","sizes":[]},"advanced_button_hover_text_color":"","button_hover_background_gradient_notice":"","button_hover_background_color":"","button_hover_background_color_stop":{"unit":"%","size":0,"sizes":[]},"button_hover_background_color_stop_tablet":{"unit":"%"},"button_hover_background_color_stop_mobile":{"unit":"%"},"button_hover_background_color_b":"#f2295b","button_hover_background_color_b_stop":{"unit":"%","size":100,"sizes":[]},"button_hover_background_color_b_stop_tablet":{"unit":"%"},"button_hover_background_color_b_stop_mobile":{"unit":"%"},"button_hover_background_gradient_type":"linear","button_hover_background_gradient_angle":{"unit":"deg","size":180,"sizes":[]},"button_hover_background_gradient_angle_tablet":{"unit":"deg"},"button_hover_background_gradient_angle_mobile":{"unit":"deg"},"button_hover_background_gradient_position":"center center","button_hover_background_image":{"url":"","id":"","size":""},"button_hover_background_image_tablet":{"url":"","id":"","size":""},"button_hover_background_image_mobile":{"url":"","id":"","size":""},"button_hover_background_position":"","button_hover_background_position_tablet":"","button_hover_background_position_mobile":"","button_hover_background_xpos":{"unit":"px","size":0,"sizes":[]},"button_hover_background_xpos_tablet":{"unit":"px","size":0,"sizes":[]},"button_hover_background_xpos_mobile":{"unit":"px","size":0,"sizes":[]},"button_hover_background_ypos":{"unit":"px","size":0,"sizes":[]},"button_hover_background_ypos_tablet":{"unit":"px","size":0,"sizes":[]},"button_hover_background_ypos_mobile":{"unit":"px","size":0,"sizes":[]},"button_hover_background_attachment":"","button_hover_background_repeat":"","button_hover_background_repeat_tablet":"","button_hover_background_repeat_mobile":"","button_hover_background_size":"","button_hover_background_size_tablet":"","button_hover_background_size_mobile":"","button_hover_background_bg_width":{"unit":"%","size":100,"sizes":[]},"button_hover_background_bg_width_tablet":{"unit":"px","size":"","sizes":[]},"button_hover_background_bg_width_mobile":{"unit":"px","size":"","sizes":[]},"button_hover_background_video_link":"","button_hover_background_video_start":"","button_hover_background_video_end":"","button_hover_background_play_once":"","button_hover_background_play_on_mobile":"","button_hover_background_privacy_mode":"","button_hover_background_video_fallback":{"url":"","id":"","size":""},"button_hover_background_slideshow_gallery":[],"button_hover_background_slideshow_loop":"yes","button_hover_background_slideshow_slide_duration":5000,"button_hover_background_slideshow_slide_transition":"fade","button_hover_background_slideshow_transition_duration":500,"button_hover_background_slideshow_background_size":"","button_hover_background_slideshow_background_size_tablet":"","button_hover_background_slideshow_background_size_mobile":"","button_hover_background_slideshow_background_position":"","button_hover_background_slideshow_background_position_tablet":"","button_hover_background_slideshow_background_position_mobile":"","button_hover_background_slideshow_lazyload":"","button_hover_background_slideshow_ken_burns":"","button_hover_background_slideshow_ken_burns_zoom_direction":"in","button_hover_border_width":{"unit":"px","top":3,"right":3,"bottom":3,"left":3,"isLinked":true},"button_hover_border_width_tablet":{"unit":"px","top":"","right":"","bottom":"","left":"","isLinked":true},"button_hover_border_width_mobile":{"unit":"px","top":"","right":"","bottom":"","left":"","isLinked":true},"button_hover_border_color":"","advanced_button_hover_radius":{"unit":"px","top":"","right":"","bottom":"","left":"","isLinked":true},"advanced_button_hover_radius_tablet":{"unit":"px","top":"","right":"","bottom":"","left":"","isLinked":true},"advanced_button_hover_radius_mobile":{"unit":"px","top":"","right":"","bottom":"","left":"","isLinked":true},"advanced_button_hover_shadow_box_shadow_type":"","advanced_button_hover_shadow_box_shadow":{"horizontal":0,"vertical":0,"blur":10,"spread":0,"color":"rgba(0,0,0,0.5)"},"advanced_button_hover_shadow_box_shadow_position":" ","hover_animation":"","advanced_button_icon_color":"","advanced_button_icon_background_background":"","advanced_button_icon_background_gradient_notice":"","advanced_button_icon_background_color":"","advanced_button_icon_background_color_stop":{"unit":"%","size":0,"sizes":[]},"advanced_button_icon_background_color_stop_tablet":{"unit":"%"},"advanced_button_icon_background_color_stop_mobile":{"unit":"%"},"advanced_button_icon_background_color_b":"#f2295b","advanced_button_icon_background_color_b_stop":{"unit":"%","size":100,"sizes":[]},"advanced_button_icon_background_color_b_stop_tablet":{"unit":"%"},"advanced_button_icon_background_color_b_stop_mobile":{"unit":"%"},"advanced_button_icon_background_gradient_type":"linear","advanced_button_icon_background_gradient_angle":{"unit":"deg","size":180,"sizes":[]},"advanced_button_icon_background_gradient_angle_tablet":{"unit":"deg"},"advanced_button_icon_background_gradient_angle_mobile":{"unit":"deg"},"advanced_button_icon_background_gradient_position":"center center","advanced_button_icon_background_image":{"url":"","id":"","size":""},"advanced_button_icon_background_image_tablet":{"url":"","id":"","size":""},"advanced_button_icon_background_image_mobile":{"url":"","id":"","size":""},"advanced_button_icon_background_position":"","advanced_button_icon_background_position_tablet":"","advanced_button_icon_background_position_mobile":"","advanced_button_icon_background_xpos":{"unit":"px","size":0,"sizes":[]},"advanced_button_icon_background_xpos_tablet":{"unit":"px","size":0,"sizes":[]},"advanced_button_icon_background_xpos_mobile":{"unit":"px","size":0,"sizes":[]},"advanced_button_icon_background_ypos":{"unit":"px","size":0,"sizes":[]},"advanced_button_icon_background_ypos_tablet":{"unit":"px","size":0,"sizes":[]},"advanced_button_icon_background_ypos_mobile":{"unit":"px","size":0,"sizes":[]},"advanced_button_icon_background_attachment":"","advanced_button_icon_background_repeat":"","advanced_button_icon_background_repeat_tablet":"","advanced_button_icon_background_repeat_mobile":"","advanced_button_icon_background_size":"","advanced_button_icon_background_size_tablet":"","advanced_button_icon_background_size_mobile":"","advanced_button_icon_background_bg_width":{"unit":"%","size":100,"sizes":[]},"advanced_button_icon_background_bg_width_tablet":{"unit":"px","size":"","sizes":[]},"advanced_button_icon_background_bg_width_mobile":{"unit":"px","size":"","sizes":[]},"advanced_button_icon_background_video_link":"","advanced_button_icon_background_video_start":"","advanced_button_icon_background_video_end":"","advanced_button_icon_background_play_once":"","advanced_button_icon_background_play_on_mobile":"","advanced_button_icon_background_privacy_mode":"","advanced_button_icon_background_video_fallback":{"url":"","id":"","size":""},"advanced_button_icon_background_slideshow_gallery":[],"advanced_button_icon_background_slideshow_loop":"yes","advanced_button_icon_background_slideshow_slide_duration":5000,"advanced_button_icon_background_slideshow_slide_transition":"fade","advanced_button_icon_background_slideshow_transition_duration":500,"advanced_button_icon_background_slideshow_background_size":"","advanced_button_icon_background_slideshow_background_size_tablet":"","advanced_button_icon_background_slideshow_background_size_mobile":"","advanced_button_icon_background_slideshow_background_position":"","advanced_button_icon_background_slideshow_background_position_tablet":"","advanced_button_icon_background_slideshow_background_position_mobile":"","advanced_button_icon_background_slideshow_lazyload":"","advanced_button_icon_background_slideshow_ken_burns":"","advanced_button_icon_background_slideshow_ken_burns_zoom_direction":"in","advanced_button_icon_border_border":"","advanced_button_icon_border_width":{"unit":"px","top":"","right":"","bottom":"","left":"","isLinked":true},"advanced_button_icon_border_width_tablet":{"unit":"px","top":"","right":"","bottom":"","left":"","isLinked":true},"advanced_button_icon_border_width_mobile":{"unit":"px","top":"","right":"","bottom":"","left":"","isLinked":true},"advanced_button_icon_border_color":"","advanced_button_icon_radius":{"unit":"px","top":"","right":"","bottom":"","left":"","isLinked":true},"advanced_button_icon_radius_tablet":{"unit":"px","top":"","right":"","bottom":"","left":"","isLinked":true},"advanced_button_icon_radius_mobile":{"unit":"px","top":"","right":"","bottom":"","left":"","isLinked":true},"advanced_button_icon_padding":{"unit":"px","top":"","right":"","bottom":"","left":"","isLinked":true},"advanced_button_icon_padding_tablet":{"unit":"px","top":"","right":"","bottom":"","left":"","isLinked":true},"advanced_button_icon_padding_mobile":{"unit":"px","top":"","right":"","bottom":"","left":"","isLinked":true},"advanced_button_icon_shadow_box_shadow_type":"","advanced_button_icon_shadow_box_shadow":{"horizontal":0,"vertical":0,"blur":10,"spread":0,"color":"rgba(0,0,0,0.5)"},"advanced_button_icon_shadow_box_shadow_position":" ","advanced_button_icon_size":{"unit":"px","size":"","sizes":[]},"advanced_button_icon_size_tablet":{"unit":"px","size":"","sizes":[]},"advanced_button_icon_size_mobile":{"unit":"px","size":"","sizes":[]},"advanced_button_hover_icon_color":"","advanced_button_icon_hover_background_background":"","advanced_button_icon_hover_background_gradient_notice":"","advanced_button_icon_hover_background_color":"","advanced_button_icon_hover_background_color_stop":{"unit":"%","size":0,"sizes":[]},"advanced_button_icon_hover_background_color_stop_tablet":{"unit":"%"},"advanced_button_icon_hover_background_color_stop_mobile":{"unit":"%"},"advanced_button_icon_hover_background_color_b":"#f2295b","advanced_button_icon_hover_background_color_b_stop":{"unit":"%","size":100,"sizes":[]},"advanced_button_icon_hover_background_color_b_stop_tablet":{"unit":"%"},"advanced_button_icon_hover_background_color_b_stop_mobile":{"unit":"%"},"advanced_button_icon_hover_background_gradient_type":"linear","advanced_button_icon_hover_background_gradient_angle":{"unit":"deg","size":180,"sizes":[]},"advanced_button_icon_hover_background_gradient_angle_tablet":{"unit":"deg"},"advanced_button_icon_hover_background_gradient_angle_mobile":{"unit":"deg"},"advanced_button_icon_hover_background_gradient_position":"center center","advanced_button_icon_hover_background_image":{"url":"","id":"","size":""},"advanced_button_icon_hover_background_image_tablet":{"url":"","id":"","size":""},"advanced_button_icon_hover_background_image_mobile":{"url":"","id":"","size":""},"advanced_button_icon_hover_background_position":"","advanced_button_icon_hover_background_position_tablet":"","advanced_button_icon_hover_background_position_mobile":"","advanced_button_icon_hover_background_xpos":{"unit":"px","size":0,"sizes":[]},"advanced_button_icon_hover_background_xpos_tablet":{"unit":"px","size":0,"sizes":[]},"advanced_button_icon_hover_background_xpos_mobile":{"unit":"px","size":0,"sizes":[]},"advanced_button_icon_hover_background_ypos":{"unit":"px","size":0,"sizes":[]},"advanced_button_icon_hover_background_ypos_tablet":{"unit":"px","size":0,"sizes":[]},"advanced_button_icon_hover_background_ypos_mobile":{"unit":"px","size":0,"sizes":[]},"advanced_button_icon_hover_background_attachment":"","advanced_button_icon_hover_background_repeat":"","advanced_button_icon_hover_background_repeat_tablet":"","advanced_button_icon_hover_background_repeat_mobile":"","advanced_button_icon_hover_background_size":"","advanced_button_icon_hover_background_size_tablet":"","advanced_button_icon_hover_background_size_mobile":"","advanced_button_icon_hover_background_bg_width":{"unit":"%","size":100,"sizes":[]},"advanced_button_icon_hover_background_bg_width_tablet":{"unit":"px","size":"","sizes":[]},"advanced_button_icon_hover_background_bg_width_mobile":{"unit":"px","size":"","sizes":[]},"advanced_button_icon_hover_background_video_link":"","advanced_button_icon_hover_background_video_start":"","advanced_button_icon_hover_background_video_end":"","advanced_button_icon_hover_background_play_once":"","advanced_button_icon_hover_background_play_on_mobile":"","advanced_button_icon_hover_background_privacy_mode":"","advanced_button_icon_hover_background_video_fallback":{"url":"","id":"","size":""},"advanced_button_icon_hover_background_slideshow_gallery":[],"advanced_button_icon_hover_background_slideshow_loop":"yes","advanced_button_icon_hover_background_slideshow_slide_duration":5000,"advanced_button_icon_hover_background_slideshow_slide_transition":"fade","advanced_button_icon_hover_background_slideshow_transition_duration":500,"advanced_button_icon_hover_background_slideshow_background_size":"","advanced_button_icon_hover_background_slideshow_background_size_tablet":"","advanced_button_icon_hover_background_slideshow_background_size_mobile":"","advanced_button_icon_hover_background_slideshow_background_position":"","advanced_button_icon_hover_background_slideshow_background_position_tablet":"","advanced_button_icon_hover_background_slideshow_background_position_mobile":"","advanced_button_icon_hover_background_slideshow_lazyload":"","advanced_button_icon_hover_background_slideshow_ken_burns":"","advanced_button_icon_hover_background_slideshow_ken_burns_zoom_direction":"in","icon_hover_border_color":"","badge_color":"","badge_background_background":"","badge_background_gradient_notice":"","badge_background_color":"","badge_background_color_stop":{"unit":"%","size":0,"sizes":[]},"badge_background_color_stop_tablet":{"unit":"%"},"badge_background_color_stop_mobile":{"unit":"%"},"badge_background_color_b":"#f2295b","badge_background_color_b_stop":{"unit":"%","size":100,"sizes":[]},"badge_background_color_b_stop_tablet":{"unit":"%"},"badge_background_color_b_stop_mobile":{"unit":"%"},"badge_background_gradient_type":"linear","badge_background_gradient_angle":{"unit":"deg","size":180,"sizes":[]},"badge_background_gradient_angle_tablet":{"unit":"deg"},"badge_background_gradient_angle_mobile":{"unit":"deg"},"badge_background_gradient_position":"center center","badge_background_image":{"url":"","id":"","size":""},"badge_background_image_tablet":{"url":"","id":"","size":""},"badge_background_image_mobile":{"url":"","id":"","size":""},"badge_background_position":"","badge_background_position_tablet":"","badge_background_position_mobile":"","badge_background_xpos":{"unit":"px","size":0,"sizes":[]},"badge_background_xpos_tablet":{"unit":"px","size":0,"sizes":[]},"badge_background_xpos_mobile":{"unit":"px","size":0,"sizes":[]},"badge_background_ypos":{"unit":"px","size":0,"sizes":[]},"badge_background_ypos_tablet":{"unit":"px","size":0,"sizes":[]},"badge_background_ypos_mobile":{"unit":"px","size":0,"sizes":[]},"badge_background_attachment":"","badge_background_repeat":"","badge_background_repeat_tablet":"","badge_background_repeat_mobile":"","badge_background_size":"","badge_background_size_tablet":"","badge_background_size_mobile":"","badge_background_bg_width":{"unit":"%","size":100,"sizes":[]},"badge_background_bg_width_tablet":{"unit":"px","size":"","sizes":[]},"badge_background_bg_width_mobile":{"unit":"px","size":"","sizes":[]},"badge_background_video_link":"","badge_background_video_start":"","badge_background_video_end":"","badge_background_play_once":"","badge_background_play_on_mobile":"","badge_background_privacy_mode":"","badge_background_video_fallback":{"url":"","id":"","size":""},"badge_background_slideshow_gallery":[],"badge_background_slideshow_loop":"yes","badge_background_slideshow_slide_duration":5000,"badge_background_slideshow_slide_transition":"fade","badge_background_slideshow_transition_duration":500,"badge_background_slideshow_background_size":"","badge_background_slideshow_background_size_tablet":"","badge_background_slideshow_background_size_mobile":"","badge_background_slideshow_background_position":"","badge_background_slideshow_background_position_tablet":"","badge_background_slideshow_background_position_mobile":"","badge_background_slideshow_lazyload":"","badge_background_slideshow_ken_burns":"","badge_background_slideshow_ken_burns_zoom_direction":"in","badge_border_border":"","badge_border_width":{"unit":"px","top":"","right":"","bottom":"","left":"","isLinked":true},"badge_border_width_tablet":{"unit":"px","top":"","right":"","bottom":"","left":"","isLinked":true},"badge_border_width_mobile":{"unit":"px","top":"","right":"","bottom":"","left":"","isLinked":true},"badge_border_color":"","badge_radius":{"unit":"px","top":"","right":"","bottom":"","left":"","isLinked":true},"badge_radius_tablet":{"unit":"px","top":"","right":"","bottom":"","left":"","isLinked":true},"badge_radius_mobile":{"unit":"px","top":"","right":"","bottom":"","left":"","isLinked":true},"badge_padding":{"unit":"px","top":"","right":"","bottom":"","left":"","isLinked":true},"badge_padding_tablet":{"unit":"px","top":"","right":"","bottom":"","left":"","isLinked":true},"badge_padding_mobile":{"unit":"px","top":"","right":"","bottom":"","left":"","isLinked":true},"badge_shadow_box_shadow_type":"","badge_shadow_box_shadow":{"horizontal":0,"vertical":0,"blur":10,"spread":0,"color":"rgba(0,0,0,0.5)"},"badge_shadow_box_shadow_position":" ","badge_typography_typography":"","badge_typography_font_family":"","badge_typography_font_size":{"unit":"px","size":"","sizes":[]},"badge_typography_font_size_tablet":{"unit":"px","size":"","sizes":[]},"badge_typography_font_size_mobile":{"unit":"px","size":"","sizes":[]},"badge_typography_font_weight":"","badge_typography_text_transform":"","badge_typography_font_style":"","badge_typography_text_decoration":"","badge_typography_line_height":{"unit":"px","size":"","sizes":[]},"badge_typography_line_height_tablet":{"unit":"em","size":"","sizes":[]},"badge_typography_line_height_mobile":{"unit":"em","size":"","sizes":[]},"badge_typography_letter_spacing":{"unit":"px","size":"","sizes":[]},"badge_typography_letter_spacing_tablet":{"unit":"px","size":"","sizes":[]},"badge_typography_letter_spacing_mobile":{"unit":"px","size":"","sizes":[]},"badge_typography_word_spacing":{"unit":"px","size":"","sizes":[]},"badge_typography_word_spacing_tablet":{"unit":"em","size":"","sizes":[]},"badge_typography_word_spacing_mobile":{"unit":"em","size":"","sizes":[]},"badge_hover_icon_color":"","badge_hover_background_background":"","badge_hover_background_gradient_notice":"","badge_hover_background_color":"","badge_hover_background_color_stop":{"unit":"%","size":0,"sizes":[]},"badge_hover_background_color_stop_tablet":{"unit":"%"},"badge_hover_background_color_stop_mobile":{"unit":"%"},"badge_hover_background_color_b":"#f2295b","badge_hover_background_color_b_stop":{"unit":"%","size":100,"sizes":[]},"badge_hover_background_color_b_stop_tablet":{"unit":"%"},"badge_hover_background_color_b_stop_mobile":{"unit":"%"},"badge_hover_background_gradient_type":"linear","badge_hover_background_gradient_angle":{"unit":"deg","size":180,"sizes":[]},"badge_hover_background_gradient_angle_tablet":{"unit":"deg"},"badge_hover_background_gradient_angle_mobile":{"unit":"deg"},"badge_hover_background_gradient_position":"center center","badge_hover_background_image":{"url":"","id":"","size":""},"badge_hover_background_image_tablet":{"url":"","id":"","size":""},"badge_hover_background_image_mobile":{"url":"","id":"","size":""},"badge_hover_background_position":"","badge_hover_background_position_tablet":"","badge_hover_background_position_mobile":"","badge_hover_background_xpos":{"unit":"px","size":0,"sizes":[]},"badge_hover_background_xpos_tablet":{"unit":"px","size":0,"sizes":[]},"badge_hover_background_xpos_mobile":{"unit":"px","size":0,"sizes":[]},"badge_hover_background_ypos":{"unit":"px","size":0,"sizes":[]},"badge_hover_background_ypos_tablet":{"unit":"px","size":0,"sizes":[]},"badge_hover_background_ypos_mobile":{"unit":"px","size":0,"sizes":[]},"badge_hover_background_attachment":"","badge_hover_background_repeat":"","badge_hover_background_repeat_tablet":"","badge_hover_background_repeat_mobile":"","badge_hover_background_size":"","badge_hover_background_size_tablet":"","badge_hover_background_size_mobile":"","badge_hover_background_bg_width":{"unit":"%","size":100,"sizes":[]},"badge_hover_background_bg_width_tablet":{"unit":"px","size":"","sizes":[]},"badge_hover_background_bg_width_mobile":{"unit":"px","size":"","sizes":[]},"badge_hover_background_video_link":"","badge_hover_background_video_start":"","badge_hover_background_video_end":"","badge_hover_background_play_once":"","badge_hover_background_play_on_mobile":"","badge_hover_background_privacy_mode":"","badge_hover_background_video_fallback":{"url":"","id":"","size":""},"badge_hover_background_slideshow_gallery":[],"badge_hover_background_slideshow_loop":"yes","badge_hover_background_slideshow_slide_duration":5000,"badge_hover_background_slideshow_slide_transition":"fade","badge_hover_background_slideshow_transition_duration":500,"badge_hover_background_slideshow_background_size":"","badge_hover_background_slideshow_background_size_tablet":"","badge_hover_background_slideshow_background_size_mobile":"","badge_hover_background_slideshow_background_position":"","badge_hover_background_slideshow_background_position_tablet":"","badge_hover_background_slideshow_background_position_mobile":"","badge_hover_background_slideshow_lazyload":"","badge_hover_background_slideshow_ken_burns":"","badge_hover_background_slideshow_ken_burns_zoom_direction":"in","badge_hover_border_color":"","_title":"","_margin":{"unit":"px","top":"","right":"","bottom":"","left":"","isLinked":true},"_margin_tablet":{"unit":"px","top":"","right":"","bottom":"","left":"","isLinked":true},"_margin_mobile":{"unit":"px","top":"","right":"","bottom":"","left":"","isLinked":true},"_padding":{"unit":"px","top":"","right":"","bottom":"","left":"","isLinked":true},"_padding_tablet":{"unit":"px","top":"","right":"","bottom":"","left":"","isLinked":true},"_padding_mobile":{"unit":"px","top":"","right":"","bottom":"","left":"","isLinked":true},"_element_width":"","_element_width_tablet":"","_element_width_mobile":"","_element_custom_width":{"unit":"%","size":"","sizes":[]},"_element_custom_width_tablet":{"unit":"px","size":"","sizes":[]},"_element_custom_width_mobile":{"unit":"px","size":"","sizes":[]},"_element_vertical_align":"","_element_vertical_align_tablet":"","_element_vertical_align_mobile":"","_position_description":"","_position":"","_offset_orientation_h":"start","_offset_x":{"unit":"px","size":0,"sizes":[]},"_offset_x_tablet":{"unit":"px","size":"","sizes":[]},"_offset_x_mobile":{"unit":"px","size":"","sizes":[]},"_offset_x_end":{"unit":"px","size":0,"sizes":[]},"_offset_x_end_tablet":{"unit":"px","size":"","sizes":[]},"_offset_x_end_mobile":{"unit":"px","size":"","sizes":[]},"_offset_orientation_v":"start","_offset_y":{"unit":"px","size":0,"sizes":[]},"_offset_y_tablet":{"unit":"px","size":"","sizes":[]},"_offset_y_mobile":{"unit":"px","size":"","sizes":[]},"_offset_y_end":{"unit":"px","size":0,"sizes":[]},"_offset_y_end_tablet":{"unit":"px","size":"","sizes":[]},"_offset_y_end_mobile":{"unit":"px","size":"","sizes":[]},"_z_index":"","_z_index_tablet":"","_z_index_mobile":"","_element_id":"","_css_classes":"","wcm_element_visibility_condition":"visible_to_everyone","wcm_element_visibility_show_plans":["wcm-all"],"wcm_element_visibility_hide_plans":["wcm-none"],"wcm_element_visibility_show_content_restricted_message":"","ep_widget_cf_confetti":"","ep_widget_cf_type":"basic","ep_widget_cf_fireworks_duration":{"unit":"px","size":"","sizes":[]},"ep_widget_cf_anim_infinite":"","ep_widget_cf_particle_count":{"unit":"px","size":"","sizes":[]},"ep_widget_cf_start_velocity":{"unit":"px","size":"","sizes":[]},"ep_widget_cf_spread":{"unit":"px","size":"","sizes":[]},"ep_widget_cf_angle":{"unit":"px","size":"","sizes":[]},"ep_widget_cf_shape_type":"basic","ep_widget_cf_scalar":{"unit":"px","size":"","sizes":[]},"ep_widget_cf_origin":"","ep_widget_cf_origin_x":{"unit":"px","size":"","sizes":[]},"ep_widget_cf_origin_y":{"unit":"px","size":"","sizes":[]},"ep_widget_cf_trigger_type":"load","ep_widget_cf_trigger_selector":"","ep_widget_cf_trigger_delay":{"unit":"px","size":3000,"sizes":[]},"ep_widget_cf_z_index":"","ep_display_conditions_enable":"","ep_display_conditions_to":"show","ep_display_conditions_relation":"all","ep_floating_effects_show":"","ep_floating_effects_translate_toggle":"","ep_floating_effects_translate_x":{"unit":"px","size":"","sizes":{"from":0,"to":0}},"ep_floating_effects_translate_y":{"unit":"px","size":"","sizes":{"from":0,"to":30}},"ep_floating_effects_translate_duration":{"unit":"px","size":1000,"sizes":[]},"ep_floating_effects_translate_delay":{"unit":"px","size":"","sizes":[]},"ep_floating_effects_rotate_toggle":"","ep_floating_effects_rotate_x":{"unit":"deg","size":"","sizes":{"from":0,"to":0}},"ep_floating_effects_rotate_y":{"unit":"deg","size":"","sizes":{"from":0,"to":0}},"ep_floating_effects_rotate_z":{"unit":"deg","size":"","sizes":{"from":0,"to":45}},"ep_floating_effects_rotate_infinite":"","ep_floating_effects_rotate_duration":{"unit":"px","size":2000,"sizes":[]},"ep_floating_effects_rotate_delay":{"unit":"px","size":"","sizes":[]},"ep_floating_effects_scale_toggle":"","ep_floating_effects_scale_x":{"unit":"px","size":"","sizes":{"from":1,"to":1.5}},"ep_floating_effects_scale_y":{"unit":"px","size":"","sizes":{"from":1,"to":1.5}},"ep_floating_effects_scale_duration":{"unit":"px","size":1000,"sizes":[]},"ep_floating_effects_scale_delay":{"unit":"px","size":"","sizes":[]},"ep_floating_effects_skew_toggle":"","ep_floating_effects_skew_x":{"unit":"px","size":"","sizes":{"from":1,"to":1.5}},"ep_floating_effects_skew_y":{"unit":"px","size":"","sizes":{"from":1,"to":1.5}},"ep_floating_effects_skew_duration":{"unit":"px","size":1000,"sizes":[]},"ep_floating_effects_skew_delay":{"unit":"px","size":"","sizes":[]},"ep_floating_effects_border_radius_toggle":"","ep_floating_effects_border_radius":{"unit":"px","size":"","sizes":{"from":0,"to":50}},"ep_floating_effects_border_radius_duration":{"unit":"px","size":1000,"sizes":[]},"ep_floating_effects_border_radius_delay":{"unit":"px","size":"","sizes":[]},"ep_floating_effects_opacity_toggle":"","ep_floating_effects_opacity_start":{"unit":"px","size":1,"sizes":[]},"ep_floating_effects_opacity_end":{"unit":"px","size":0,"sizes":[]},"ep_floating_effects_opacity_duration":{"unit":"px","size":1000,"sizes":[]},"ep_floating_effects_easing":"easeInOutQuad","ep_parallax_effects_show":"","ep_parallax_effects_x":"","ep_parallax_effects_x_start":{"unit":"px","size":"","sizes":[]},"ep_parallax_effects_x_end":{"unit":"px","size":"","sizes":[]},"ep_parallax_effects_x_custom_show":"","ep_parallax_effects_x_custom_value":"","ep_parallax_effects_y":"","ep_parallax_effects_y_start":{"unit":"px","size":50,"sizes":[]},"ep_parallax_effects_y_end":{"unit":"px","size":0,"sizes":[]},"ep_parallax_effects_y_custom_show":"","ep_parallax_effects_y_custom_value":"","ep_parallax_effects_opacity_toggole":"","ep_parallax_effects_opacity":"","ep_parallax_effects_opacity_custom_show":"","ep_parallax_effects_opacity_custom_value":"","ep_parallax_effects_blur":"","ep_parallax_effects_blur_start":{"unit":"px","size":"","sizes":[]},"ep_parallax_effects_blur_end":{"unit":"px","size":"","sizes":[]},"ep_parallax_effects_rotate":"","ep_parallax_effects_rotate_start":{"unit":"px","size":"","sizes":[]},"ep_parallax_effects_rotate_end":{"unit":"px","size":"","sizes":[]},"ep_parallax_effects_scale":"","ep_parallax_effects_scale_start":{"unit":"px","size":1,"sizes":[]},"ep_parallax_effects_scale_end":{"unit":"px","size":1,"sizes":[]},"ep_parallax_effects_hue":"","ep_parallax_effects_hue_value":{"unit":"px","size":"","sizes":[]},"ep_parallax_effects_sepia":"","ep_parallax_effects_sepia_value":{"unit":"px","size":1,"sizes":[]},"ep_parallax_effects_easing":"","ep_parallax_effects_easing_value":{"unit":"px","size":1,"sizes":[]},"ep_parallax_effects_transition":"","ep_parallax_effects_viewport":"","ep_parallax_effects_viewport_start":"","ep_parallax_effects_viewport_end":"","ep_parallax_effects_media_query":"","ep_parallax_effects_target":"self","motion_fx_motion_fx_scrolling":"","motion_fx_translateY_effect":"","motion_fx_translateY_direction":"","motion_fx_translateY_speed":{"unit":"px","size":4,"sizes":[]},"motion_fx_translateY_affectedRange":{"unit":"%","size":"","sizes":{"start":0,"end":100}},"motion_fx_translateX_effect":"","motion_fx_translateX_direction":"","motion_fx_translateX_speed":{"unit":"px","size":4,"sizes":[]},"motion_fx_translateX_affectedRange":{"unit":"%","size":"","sizes":{"start":0,"end":100}},"motion_fx_opacity_effect":"","motion_fx_opacity_direction":"out-in","motion_fx_opacity_level":{"unit":"px","size":10,"sizes":[]},"motion_fx_opacity_range":{"unit":"%","size":"","sizes":{"start":20,"end":80}},"motion_fx_blur_effect":"","motion_fx_blur_direction":"out-in","motion_fx_blur_level":{"unit":"px","size":7,"sizes":[]},"motion_fx_blur_range":{"unit":"%","size":"","sizes":{"start":20,"end":80}},"motion_fx_rotateZ_effect":"","motion_fx_rotateZ_direction":"","motion_fx_rotateZ_speed":{"unit":"px","size":1,"sizes":[]},"motion_fx_rotateZ_affectedRange":{"unit":"%","size":"","sizes":{"start":0,"end":100}},"motion_fx_scale_effect":"","motion_fx_scale_direction":"out-in","motion_fx_scale_speed":{"unit":"px","size":4,"sizes":[]},"motion_fx_scale_range":{"unit":"%","size":"","sizes":{"start":20,"end":80}},"motion_fx_transform_origin_x":"center","motion_fx_transform_origin_y":"center","motion_fx_devices":["desktop","tablet","mobile"],"motion_fx_range":"","motion_fx_motion_fx_mouse":"","motion_fx_mouseTrack_effect":"","motion_fx_mouseTrack_direction":"","motion_fx_mouseTrack_speed":{"unit":"px","size":1,"sizes":[]},"motion_fx_tilt_effect":"","motion_fx_tilt_direction":"","motion_fx_tilt_speed":{"unit":"px","size":4,"sizes":[]},"sticky":"","sticky_on":["desktop","tablet","mobile"],"sticky_offset":0,"sticky_offset_tablet":"","sticky_offset_mobile":"","sticky_effects_offset":0,"sticky_effects_offset_tablet":"","sticky_effects_offset_mobile":"","sticky_parent":"","_animation":"","_animation_tablet":"","_animation_mobile":"","animation_duration":"","_animation_delay":"","_transform_rotate_popover":"","_transform_rotateZ_effect":{"unit":"px","size":"","sizes":[]},"_transform_rotateZ_effect_tablet":{"unit":"deg","size":"","sizes":[]},"_transform_rotateZ_effect_mobile":{"unit":"deg","size":"","sizes":[]},"_transform_rotate_3d":"","_transform_rotateX_effect":{"unit":"px","size":"","sizes":[]},"_transform_rotateX_effect_tablet":{"unit":"deg","size":"","sizes":[]},"_transform_rotateX_effect_mobile":{"unit":"deg","size":"","sizes":[]},"_transform_rotateY_effect":{"unit":"px","size":"","sizes":[]},"_transform_rotateY_effect_tablet":{"unit":"deg","size":"","sizes":[]},"_transform_rotateY_effect_mobile":{"unit":"deg","size":"","sizes":[]},"_transform_perspective_effect":{"unit":"px","size":"","sizes":[]},"_transform_perspective_effect_tablet":{"unit":"px","size":"","sizes":[]},"_transform_perspective_effect_mobile":{"unit":"px","size":"","sizes":[]},"_transform_translate_popover":"","_transform_translateX_effect":{"unit":"px","size":"","sizes":[]},"_transform_translateX_effect_tablet":{"unit":"px","size":"","sizes":[]},"_transform_translateX_effect_mobile":{"unit":"px","size":"","sizes":[]},"_transform_translateY_effect":{"unit":"px","size":"","sizes":[]},"_transform_translateY_effect_tablet":{"unit":"px","size":"","sizes":[]},"_transform_translateY_effect_mobile":{"unit":"px","size":"","sizes":[]},"_transform_scale_popover":"","_transform_keep_proportions":"yes","_transform_scale_effect":{"unit":"px","size":"","sizes":[]},"_transform_scale_effect_tablet":{"unit":"px","size":"","sizes":[]},"_transform_scale_effect_mobile":{"unit":"px","size":"","sizes":[]},"_transform_scaleX_effect":{"unit":"px","size":"","sizes":[]},"_transform_scaleX_effect_tablet":{"unit":"px","size":"","sizes":[]},"_transform_scaleX_effect_mobile":{"unit":"px","size":"","sizes":[]},"_transform_scaleY_effect":{"unit":"px","size":"","sizes":[]},"_transform_scaleY_effect_tablet":{"unit":"px","size":"","sizes":[]},"_transform_scaleY_effect_mobile":{"unit":"px","size":"","sizes":[]},"_transform_skew_popover":"","_transform_skewX_effect":{"unit":"px","size":"","sizes":[]},"_transform_skewX_effect_tablet":{"unit":"deg","size":"","sizes":[]},"_transform_skewX_effect_mobile":{"unit":"deg","size":"","sizes":[]},"_transform_skewY_effect":{"unit":"px","size":"","sizes":[]},"_transform_skewY_effect_tablet":{"unit":"deg","size":"","sizes":[]},"_transform_skewY_effect_mobile":{"unit":"deg","size":"","sizes":[]},"_transform_flipX_effect":"","_transform_flipY_effect":"","_transform_rotate_popover_hover":"","_transform_rotateZ_effect_hover":{"unit":"px","size":"","sizes":[]},"_transform_rotateZ_effect_hover_tablet":{"unit":"deg","size":"","sizes":[]},"_transform_rotateZ_effect_hover_mobile":{"unit":"deg","size":"","sizes":[]},"_transform_rotate_3d_hover":"","_transform_rotateX_effect_hover":{"unit":"px","size":"","sizes":[]},"_transform_rotateX_effect_hover_tablet":{"unit":"deg","size":"","sizes":[]},"_transform_rotateX_effect_hover_mobile":{"unit":"deg","size":"","sizes":[]},"_transform_rotateY_effect_hover":{"unit":"px","size":"","sizes":[]},"_transform_rotateY_effect_hover_tablet":{"unit":"deg","size":"","sizes":[]},"_transform_rotateY_effect_hover_mobile":{"unit":"deg","size":"","sizes":[]},"_transform_perspective_effect_hover":{"unit":"px","size":"","sizes":[]},"_transform_perspective_effect_hover_tablet":{"unit":"px","size":"","sizes":[]},"_transform_perspective_effect_hover_mobile":{"unit":"px","size":"","sizes":[]},"_transform_translate_popover_hover":"","_transform_translateX_effect_hover":{"unit":"px","size":"","sizes":[]},"_transform_translateX_effect_hover_tablet":{"unit":"px","size":"","sizes":[]},"_transform_translateX_effect_hover_mobile":{"unit":"px","size":"","sizes":[]},"_transform_translateY_effect_hover":{"unit":"px","size":"","sizes":[]},"_transform_translateY_effect_hover_tablet":{"unit":"px","size":"","sizes":[]},"_transform_translateY_effect_hover_mobile":{"unit":"px","size":"","sizes":[]},"_transform_scale_popover_hover":"","_transform_keep_proportions_hover":"yes","_transform_scale_effect_hover":{"unit":"px","size":"","sizes":[]},"_transform_scale_effect_hover_tablet":{"unit":"px","size":"","sizes":[]},"_transform_scale_effect_hover_mobile":{"unit":"px","size":"","sizes":[]},"_transform_scaleX_effect_hover":{"unit":"px","size":"","sizes":[]},"_transform_scaleX_effect_hover_tablet":{"unit":"px","size":"","sizes":[]},"_transform_scaleX_effect_hover_mobile":{"unit":"px","size":"","sizes":[]},"_transform_scaleY_effect_hover":{"unit":"px","size":"","sizes":[]},"_transform_scaleY_effect_hover_tablet":{"unit":"px","size":"","sizes":[]},"_transform_scaleY_effect_hover_mobile":{"unit":"px","size":"","sizes":[]},"_transform_skew_popover_hover":"","_transform_skewX_effect_hover":{"unit":"px","size":"","sizes":[]},"_transform_skewX_effect_hover_tablet":{"unit":"deg","size":"","sizes":[]},"_transform_skewX_effect_hover_mobile":{"unit":"deg","size":"","sizes":[]},"_transform_skewY_effect_hover":{"unit":"px","size":"","sizes":[]},"_transform_skewY_effect_hover_tablet":{"unit":"deg","size":"","sizes":[]},"_transform_skewY_effect_hover_mobile":{"unit":"deg","size":"","sizes":[]},"_transform_flipX_effect_hover":"","_transform_flipY_effect_hover":"","_transform_transition_hover":{"unit":"px","size":"","sizes":[]},"motion_fx_transform_x_anchor_point":"","motion_fx_transform_x_anchor_point_tablet":"","motion_fx_transform_x_anchor_point_mobile":"","motion_fx_transform_y_anchor_point":"","motion_fx_transform_y_anchor_point_tablet":"","motion_fx_transform_y_anchor_point_mobile":"","_background_background":"","_background_gradient_notice":"","_background_color":"","_background_color_stop":{"unit":"%","size":0,"sizes":[]},"_background_color_stop_tablet":{"unit":"%"},"_background_color_stop_mobile":{"unit":"%"},"_background_color_b":"#f2295b","_background_color_b_stop":{"unit":"%","size":100,"sizes":[]},"_background_color_b_stop_tablet":{"unit":"%"},"_background_color_b_stop_mobile":{"unit":"%"},"_background_gradient_type":"linear","_background_gradient_angle":{"unit":"deg","size":180,"sizes":[]},"_background_gradient_angle_tablet":{"unit":"deg"},"_background_gradient_angle_mobile":{"unit":"deg"},"_background_gradient_position":"center center","_background_image":{"url":"","id":"","size":""},"_background_image_tablet":{"url":"","id":"","size":""},"_background_image_mobile":{"url":"","id":"","size":""},"_background_position":"","_background_position_tablet":"","_background_position_mobile":"","_background_xpos":{"unit":"px","size":0,"sizes":[]},"_background_xpos_tablet":{"unit":"px","size":0,"sizes":[]},"_background_xpos_mobile":{"unit":"px","size":0,"sizes":[]},"_background_ypos":{"unit":"px","size":0,"sizes":[]},"_background_ypos_tablet":{"unit":"px","size":0,"sizes":[]},"_background_ypos_mobile":{"unit":"px","size":0,"sizes":[]},"_background_attachment":"","_background_repeat":"","_background_repeat_tablet":"","_background_repeat_mobile":"","_background_size":"","_background_size_tablet":"","_background_size_mobile":"","_background_bg_width":{"unit":"%","size":100,"sizes":[]},"_background_bg_width_tablet":{"unit":"px","size":"","sizes":[]},"_background_bg_width_mobile":{"unit":"px","size":"","sizes":[]},"_background_video_link":"","_background_video_start":"","_background_video_end":"","_background_play_once":"","_background_play_on_mobile":"","_background_privacy_mode":"","_background_video_fallback":{"url":"","id":"","size":""},"_background_slideshow_gallery":[],"_background_slideshow_loop":"yes","_background_slideshow_slide_duration":5000,"_background_slideshow_slide_transition":"fade","_background_slideshow_transition_duration":500,"_background_slideshow_background_size":"","_background_slideshow_background_size_tablet":"","_background_slideshow_background_size_mobile":"","_background_slideshow_background_position":"","_background_slideshow_background_position_tablet":"","_background_slideshow_background_position_mobile":"","_background_slideshow_lazyload":"","_background_slideshow_ken_burns":"","_background_slideshow_ken_burns_zoom_direction":"in","_background_hover_background":"","_background_hover_gradient_notice":"","_background_hover_color":"","_background_hover_color_stop":{"unit":"%","size":0,"sizes":[]},"_background_hover_color_stop_tablet":{"unit":"%"},"_background_hover_color_stop_mobile":{"unit":"%"},"_background_hover_color_b":"#f2295b","_background_hover_color_b_stop":{"unit":"%","size":100,"sizes":[]},"_background_hover_color_b_stop_tablet":{"unit":"%"},"_background_hover_color_b_stop_mobile":{"unit":"%"},"_background_hover_gradient_type":"linear","_background_hover_gradient_angle":{"unit":"deg","size":180,"sizes":[]},"_background_hover_gradient_angle_tablet":{"unit":"deg"},"_background_hover_gradient_angle_mobile":{"unit":"deg"},"_background_hover_gradient_position":"center center","_background_hover_image":{"url":"","id":"","size":""},"_background_hover_image_tablet":{"url":"","id":"","size":""},"_background_hover_image_mobile":{"url":"","id":"","size":""},"_background_hover_position":"","_background_hover_position_tablet":"","_background_hover_position_mobile":"","_background_hover_xpos":{"unit":"px","size":0,"sizes":[]},"_background_hover_xpos_tablet":{"unit":"px","size":0,"sizes":[]},"_background_hover_xpos_mobile":{"unit":"px","size":0,"sizes":[]},"_background_hover_ypos":{"unit":"px","size":0,"sizes":[]},"_background_hover_ypos_tablet":{"unit":"px","size":0,"sizes":[]},"_background_hover_ypos_mobile":{"unit":"px","size":0,"sizes":[]},"_background_hover_attachment":"","_background_hover_repeat":"","_background_hover_repeat_tablet":"","_background_hover_repeat_mobile":"","_background_hover_size":"","_background_hover_size_tablet":"","_background_hover_size_mobile":"","_background_hover_bg_width":{"unit":"%","size":100,"sizes":[]},"_background_hover_bg_width_tablet":{"unit":"px","size":"","sizes":[]},"_background_hover_bg_width_mobile":{"unit":"px","size":"","sizes":[]},"_background_hover_video_link":"","_background_hover_video_start":"","_background_hover_video_end":"","_background_hover_play_once":"","_background_hover_play_on_mobile":"","_background_hover_privacy_mode":"","_background_hover_video_fallback":{"url":"","id":"","size":""},"_background_hover_slideshow_gallery":[],"_background_hover_slideshow_loop":"yes","_background_hover_slideshow_slide_duration":5000,"_background_hover_slideshow_slide_transition":"fade","_background_hover_slideshow_transition_duration":500,"_background_hover_slideshow_background_size":"","_background_hover_slideshow_background_size_tablet":"","_background_hover_slideshow_background_size_mobile":"","_background_hover_slideshow_background_position":"","_background_hover_slideshow_background_position_tablet":"","_background_hover_slideshow_background_position_mobile":"","_background_hover_slideshow_lazyload":"","_background_hover_slideshow_ken_burns":"","_background_hover_slideshow_ken_burns_zoom_direction":"in","_background_hover_transition":{"unit":"px","size":"","sizes":[]},"ep_background_overlay_background":"","ep_background_overlay_gradient_notice":"","ep_background_overlay_color":"","ep_background_overlay_color_stop":{"unit":"%","size":0,"sizes":[]},"ep_background_overlay_color_stop_tablet":{"unit":"%"},"ep_background_overlay_color_stop_mobile":{"unit":"%"},"ep_background_overlay_color_b":"#f2295b","ep_background_overlay_color_b_stop":{"unit":"%","size":100,"sizes":[]},"ep_background_overlay_color_b_stop_tablet":{"unit":"%"},"ep_background_overlay_color_b_stop_mobile":{"unit":"%"},"ep_background_overlay_gradient_type":"linear","ep_background_overlay_gradient_angle":{"unit":"deg","size":180,"sizes":[]},"ep_background_overlay_gradient_angle_tablet":{"unit":"deg"},"ep_background_overlay_gradient_angle_mobile":{"unit":"deg"},"ep_background_overlay_gradient_position":"center center","ep_background_overlay_image":{"url":"","id":"","size":""},"ep_background_overlay_image_tablet":{"url":"","id":"","size":""},"ep_background_overlay_image_mobile":{"url":"","id":"","size":""},"ep_background_overlay_position":"","ep_background_overlay_position_tablet":"","ep_background_overlay_position_mobile":"","ep_background_overlay_xpos":{"unit":"px","size":0,"sizes":[]},"ep_background_overlay_xpos_tablet":{"unit":"px","size":0,"sizes":[]},"ep_background_overlay_xpos_mobile":{"unit":"px","size":0,"sizes":[]},"ep_background_overlay_ypos":{"unit":"px","size":0,"sizes":[]},"ep_background_overlay_ypos_tablet":{"unit":"px","size":0,"sizes":[]},"ep_background_overlay_ypos_mobile":{"unit":"px","size":0,"sizes":[]},"ep_background_overlay_attachment":"","ep_background_overlay_repeat":"","ep_background_overlay_repeat_tablet":"","ep_background_overlay_repeat_mobile":"","ep_background_overlay_size":"","ep_background_overlay_size_tablet":"","ep_background_overlay_size_mobile":"","ep_background_overlay_bg_width":{"unit":"%","size":100,"sizes":[]},"ep_background_overlay_bg_width_tablet":{"unit":"px","size":"","sizes":[]},"ep_background_overlay_bg_width_mobile":{"unit":"px","size":"","sizes":[]},"ep_background_overlay_video_link":"","ep_background_overlay_video_start":"","ep_background_overlay_video_end":"","ep_background_overlay_play_once":"","ep_background_overlay_play_on_mobile":"","ep_background_overlay_privacy_mode":"","ep_background_overlay_video_fallback":{"url":"","id":"","size":""},"ep_background_overlay_slideshow_gallery":[],"ep_background_overlay_slideshow_loop":"yes","ep_background_overlay_slideshow_slide_duration":5000,"ep_background_overlay_slideshow_slide_transition":"fade","ep_background_overlay_slideshow_transition_duration":500,"ep_background_overlay_slideshow_background_size":"","ep_background_overlay_slideshow_background_size_tablet":"","ep_background_overlay_slideshow_background_size_mobile":"","ep_background_overlay_slideshow_background_position":"","ep_background_overlay_slideshow_background_position_tablet":"","ep_background_overlay_slideshow_background_position_mobile":"","ep_background_overlay_slideshow_lazyload":"","ep_background_overlay_slideshow_ken_burns":"","ep_background_overlay_slideshow_ken_burns_zoom_direction":"in","ep_background_overlay_opacity":{"unit":"px","size":0.5,"sizes":[]},"ep_css_filters_css_filter":"","ep_css_filters_blur":{"unit":"px","size":0,"sizes":[]},"ep_css_filters_brightness":{"unit":"px","size":100,"sizes":[]},"ep_css_filters_contrast":{"unit":"px","size":100,"sizes":[]},"ep_css_filters_saturate":{"unit":"px","size":100,"sizes":[]},"ep_css_filters_hue":{"unit":"px","size":0,"sizes":[]},"ep_overlay_blend_mode":"","ep_background_overlay_radius":{"unit":"px","top":"","right":"","bottom":"","left":"","isLinked":true},"ep_background_overlay_radius_tablet":{"unit":"px","top":"","right":"","bottom":"","left":"","isLinked":true},"ep_background_overlay_radius_mobile":{"unit":"px","top":"","right":"","bottom":"","left":"","isLinked":true},"ep_background_overlay_hover_background":"","ep_background_overlay_hover_gradient_notice":"","ep_background_overlay_hover_color":"","ep_background_overlay_hover_color_stop":{"unit":"%","size":0,"sizes":[]},"ep_background_overlay_hover_color_stop_tablet":{"unit":"%"},"ep_background_overlay_hover_color_stop_mobile":{"unit":"%"},"ep_background_overlay_hover_color_b":"#f2295b","ep_background_overlay_hover_color_b_stop":{"unit":"%","size":100,"sizes":[]},"ep_background_overlay_hover_color_b_stop_tablet":{"unit":"%"},"ep_background_overlay_hover_color_b_stop_mobile":{"unit":"%"},"ep_background_overlay_hover_gradient_type":"linear","ep_background_overlay_hover_gradient_angle":{"unit":"deg","size":180,"sizes":[]},"ep_background_overlay_hover_gradient_angle_tablet":{"unit":"deg"},"ep_background_overlay_hover_gradient_angle_mobile":{"unit":"deg"},"ep_background_overlay_hover_gradient_position":"center center","ep_background_overlay_hover_image":{"url":"","id":"","size":""},"ep_background_overlay_hover_image_tablet":{"url":"","id":"","size":""},"ep_background_overlay_hover_image_mobile":{"url":"","id":"","size":""},"ep_background_overlay_hover_position":"","ep_background_overlay_hover_position_tablet":"","ep_background_overlay_hover_position_mobile":"","ep_background_overlay_hover_xpos":{"unit":"px","size":0,"sizes":[]},"ep_background_overlay_hover_xpos_tablet":{"unit":"px","size":0,"sizes":[]},"ep_background_overlay_hover_xpos_mobile":{"unit":"px","size":0,"sizes":[]},"ep_background_overlay_hover_ypos":{"unit":"px","size":0,"sizes":[]},"ep_background_overlay_hover_ypos_tablet":{"unit":"px","size":0,"sizes":[]},"ep_background_overlay_hover_ypos_mobile":{"unit":"px","size":0,"sizes":[]},"ep_background_overlay_hover_attachment":"","ep_background_overlay_hover_repeat":"","ep_background_overlay_hover_repeat_tablet":"","ep_background_overlay_hover_repeat_mobile":"","ep_background_overlay_hover_size":"","ep_background_overlay_hover_size_tablet":"","ep_background_overlay_hover_size_mobile":"","ep_background_overlay_hover_bg_width":{"unit":"%","size":100,"sizes":[]},"ep_background_overlay_hover_bg_width_tablet":{"unit":"px","size":"","sizes":[]},"ep_background_overlay_hover_bg_width_mobile":{"unit":"px","size":"","sizes":[]},"ep_background_overlay_hover_video_link":"","ep_background_overlay_hover_video_start":"","ep_background_overlay_hover_video_end":"","ep_background_overlay_hover_play_once":"","ep_background_overlay_hover_play_on_mobile":"","ep_background_overlay_hover_privacy_mode":"","ep_background_overlay_hover_video_fallback":{"url":"","id":"","size":""},"ep_background_overlay_hover_slideshow_gallery":[],"ep_background_overlay_hover_slideshow_loop":"yes","ep_background_overlay_hover_slideshow_slide_duration":5000,"ep_background_overlay_hover_slideshow_slide_transition":"fade","ep_background_overlay_hover_slideshow_transition_duration":500,"ep_background_overlay_hover_slideshow_background_size":"","ep_background_overlay_hover_slideshow_background_size_tablet":"","ep_background_overlay_hover_slideshow_background_size_mobile":"","ep_background_overlay_hover_slideshow_background_position":"","ep_background_overlay_hover_slideshow_background_position_tablet":"","ep_background_overlay_hover_slideshow_background_position_mobile":"","ep_background_overlay_hover_slideshow_lazyload":"","ep_background_overlay_hover_slideshow_ken_burns":"","ep_background_overlay_hover_slideshow_ken_burns_zoom_direction":"in","ep_background_overlay_hover_opacity":{"unit":"px","size":0.5,"sizes":[]},"ep_css_filters_hover_css_filter":"","ep_css_filters_hover_blur":{"unit":"px","size":0,"sizes":[]},"ep_css_filters_hover_brightness":{"unit":"px","size":100,"sizes":[]},"ep_css_filters_hover_contrast":{"unit":"px","size":100,"sizes":[]},"ep_css_filters_hover_saturate":{"unit":"px","size":100,"sizes":[]},"ep_css_filters_hover_hue":{"unit":"px","size":0,"sizes":[]},"ep_background_overlay_hover_transition_duration":{"unit":"px","size":0.3,"sizes":[]},"ep_background_overlay_hover_radius":{"unit":"px","top":"","right":"","bottom":"","left":"","isLinked":true},"ep_background_overlay_hover_radius_tablet":{"unit":"px","top":"","right":"","bottom":"","left":"","isLinked":true},"ep_background_overlay_hover_radius_mobile":{"unit":"px","top":"","right":"","bottom":"","left":"","isLinked":true},"ep_background_overlay_margin":{"unit":"px","top":"","right":"","bottom":"","left":"","isLinked":true},"ep_background_overlay_margin_tablet":{"unit":"px","top":"","right":"","bottom":"","left":"","isLinked":true},"ep_background_overlay_margin_mobile":{"unit":"px","top":"","right":"","bottom":"","left":"","isLinked":true},"ep_background_overlay_zindex":"","ep_background_overlay_position_relative":"","ep_background_overlay_widget_zindex":"-1","_border_border":"","_border_width":{"unit":"px","top":"","right":"","bottom":"","left":"","isLinked":true},"_border_width_tablet":{"unit":"px","top":"","right":"","bottom":"","left":"","isLinked":true},"_border_width_mobile":{"unit":"px","top":"","right":"","bottom":"","left":"","isLinked":true},"_border_color":"","_border_radius":{"unit":"px","top":"","right":"","bottom":"","left":"","isLinked":true},"_border_radius_tablet":{"unit":"px","top":"","right":"","bottom":"","left":"","isLinked":true},"_border_radius_mobile":{"unit":"px","top":"","right":"","bottom":"","left":"","isLinked":true},"_box_shadow_box_shadow_type":"","_box_shadow_box_shadow":{"horizontal":0,"vertical":0,"blur":10,"spread":0,"color":"rgba(0,0,0,0.5)"},"_box_shadow_box_shadow_position":" ","_border_hover_border":"","_border_hover_width":{"unit":"px","top":"","right":"","bottom":"","left":"","isLinked":true},"_border_hover_width_tablet":{"unit":"px","top":"","right":"","bottom":"","left":"","isLinked":true},"_border_hover_width_mobile":{"unit":"px","top":"","right":"","bottom":"","left":"","isLinked":true},"_border_hover_color":"","_border_radius_hover":{"unit":"px","top":"","right":"","bottom":"","left":"","isLinked":true},"_border_radius_hover_tablet":{"unit":"px","top":"","right":"","bottom":"","left":"","isLinked":true},"_border_radius_hover_mobile":{"unit":"px","top":"","right":"","bottom":"","left":"","isLinked":true},"_box_shadow_hover_box_shadow_type":"","_box_shadow_hover_box_shadow":{"horizontal":0,"vertical":0,"blur":10,"spread":0,"color":"rgba(0,0,0,0.5)"},"_box_shadow_hover_box_shadow_position":" ","_border_hover_transition":{"unit":"px","size":"","sizes":[]},"_mask_switch":"","_mask_shape":"circle","_mask_image":{"url":"","id":"","size":""},"_mask_notice":"","_mask_size":"contain","_mask_size_tablet":"","_mask_size_mobile":"","_mask_size_scale":{"unit":"%","size":100,"sizes":[]},"_mask_size_scale_tablet":{"unit":"px","size":"","sizes":[]},"_mask_size_scale_mobile":{"unit":"px","size":"","sizes":[]},"_mask_position":"center center","_mask_position_tablet":"","_mask_position_mobile":"","_mask_position_x":{"unit":"%","size":0,"sizes":[]},"_mask_position_x_tablet":{"unit":"px","size":"","sizes":[]},"_mask_position_x_mobile":{"unit":"px","size":"","sizes":[]},"_mask_position_y":{"unit":"%","size":0,"sizes":[]},"_mask_position_y_tablet":{"unit":"px","size":"","sizes":[]},"_mask_position_y_mobile":{"unit":"px","size":"","sizes":[]},"_mask_repeat":"no-repeat","_mask_repeat_tablet":"","_mask_repeat_mobile":"","hide_desktop":"","hide_tablet":"","hide_mobile":"","_attributes":"","custom_css":""},"defaultEditSettings":{"defaultEditRoute":"content"},"elements":[],"widgetType":"bdt-advanced-button","editSettings":{"defaultEditRoute":"content"},"htmlCache":""}]}](https://consultorsalud.com/wp-content/uploads/2024/03/1080x1080_portal_central-de-mezclas_01-1.jpg)
